DNA: Estrutura e Replicação
Figuras Legendadas
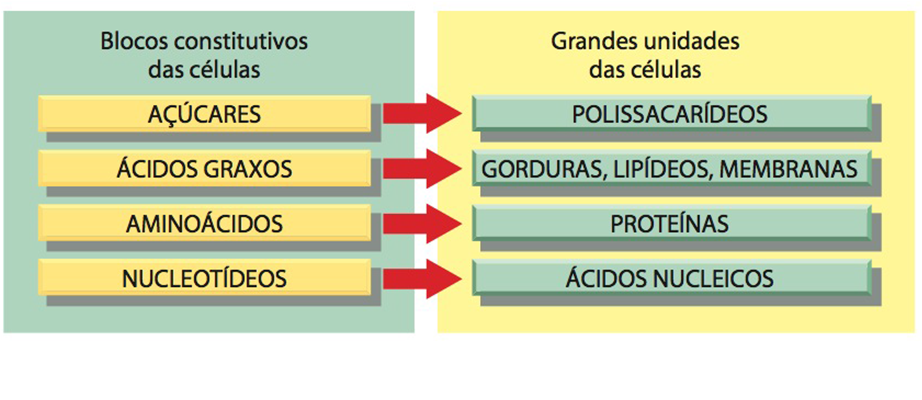
Fig. 1 Moléculas da Matéria Viva. Na imagem estão listadas as principais Moléculas da Matéria Viva, que compõe a estrutura e executam todas as funções dos seres vivos. Os açúcares são as subunidades constituintes dos polissacarídeos e desempenham função estrutural e armazenadora de energia. Os ácidos graxos abrangem as gorduras e lipídeos, são responsáveis por armazenar energia, isolar e formar membranas semipermeáveis. Os aminoácidos através de ligações peptídicas formam proteínas que exercem grande parte das funções das células: função estrutural, enzimática, sinalizadora etc. Os nucleotídeos são compostos que transportam muita energia e portanto auxiliam nos processos metabólicos, especialmente as biossínteses, na maioria das células. Atuam também como sinais químicos, respondendo assim a hormônios e outros estímulos extracelulares. Além disso, são também componentes estruturais de cofatores enzimáticos, intermediários metabólicos e ácidos nucleicos (DNA e RNA).
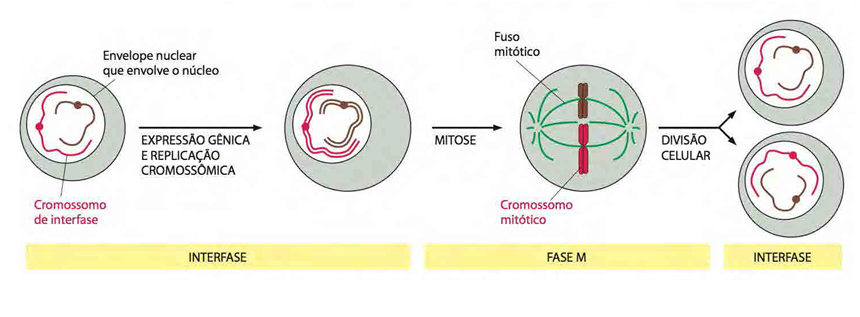
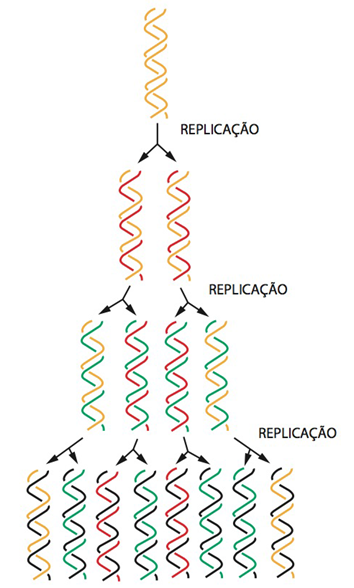
Fig. 2 Divisão Celular. A imagem representa a mitose, um tipo de divisão celular em que as células resultantes possuem exatamente o mesmo material genético que a célula inicial. Uma célula n produz duas células n, uma célula 2n produz duas células 2n etc, portanto trata-se de uma divisão equacional. Como observamos, interfase é o período que precede qualquer divisão celular, sendo de intensa atividade metabólica e portanto a principal atividade da célula, que antes de se dividir, duplica todo seu material genético presente no núcleo. Nesse período, há a preparação para a divisão celular, que envolve a duplicação da cromatina (DNA condensado através de proteínas específicas denominadas histonas) . As duas cópias de cada cromossomo permanecem juntas, unidas por um centrômero, constituindo duas cromátides de um mesmo cromossomo. Na fase M as cromátides irmãs são segregadas através da interação com microtúbulos, proteínas motoras e centrossomos, distribuindo igualmente o material genético para as duas células filhas.
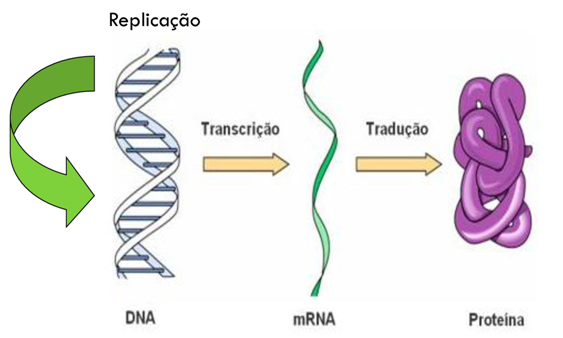
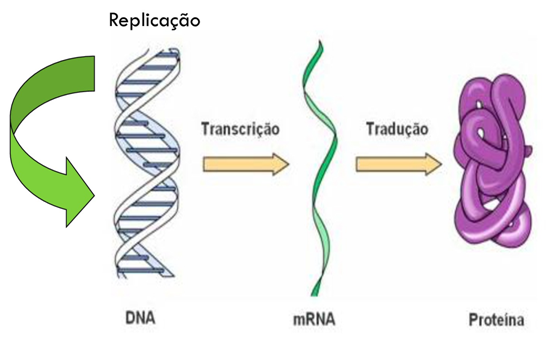
Fig. 3 Dogma Central da Biologia Molecular. O Dogma Central da Biologia Molecular foi postulado por Francis Crick em 1958. Ele explica de maneira ilustrativa, através de um esquema, como ocorre o fluxo de informações do código genético. Inicialmente observamos a Replicação, em que o DNA é duplicado, e como vimos na imagem anterior este processo ocorre somente durante a divisão celular. A Transcrição representa a passagem das informações presentes em um gene (fragmento do DNA) para um RNA, para que o mesmo carregue a informação necessária para a produção de uma proteína, ou seja, é o passo inicial para expressão genética (é uma ação constante para célula assim como a tradução) . A Tradução é o processo de codificação dos nucleotídeos presentes no RNA em aminoácidos correspondentes, a cada três nucleotídeos é formado um códon que expressa um aminoácido específico. A união de vários aminoácidos através de ligações peptídicas, como vimos na figura 1, produz como resultado uma proteína.
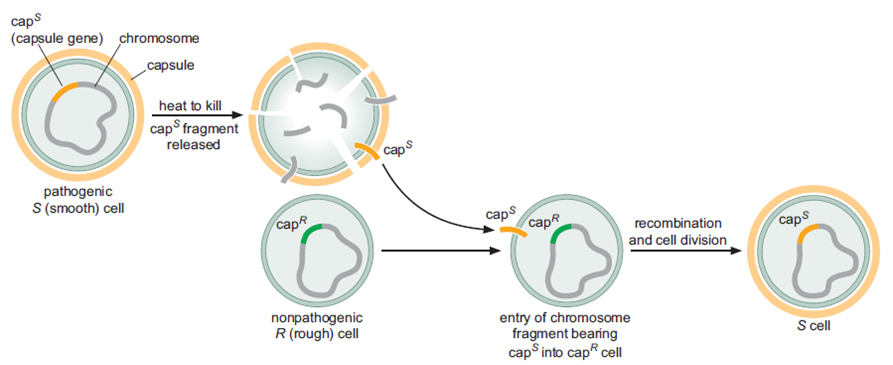
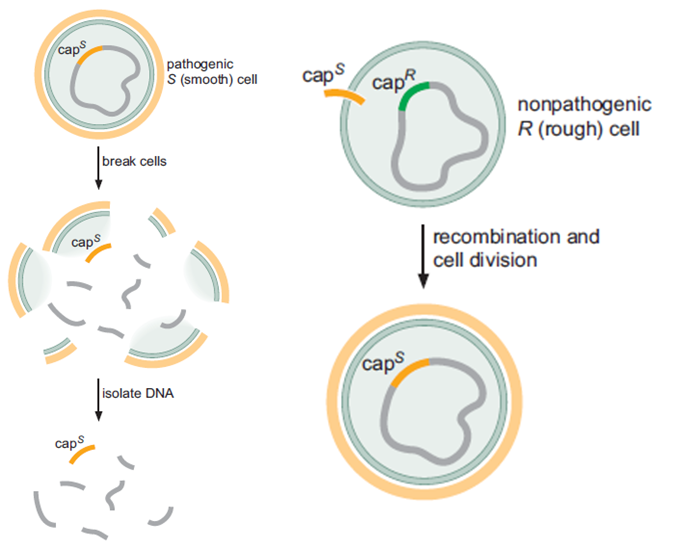
Fig. 4 Experimento de Griffith (1928) e Principio Transformante. Este experimento foi utilizado para comprovar a associação direta entre o material genético das células com suas características e hereditariedade. Griffith utilizou duas bactérias parecidas, porém não iguais: CAP S (com cápsula; patogênica) e CAP R (sem capsula; não patogênica). Inicialmente ao introduzir a bactéria CAP S viva em um rato, após um período, o mesmo foi à óbito. E introduzindo a bactéria CAP R viva no rato, o mesmo permanece vivo. Ao aquecer a bactéria CAP S e promover a sua morte, Griffith percebeu que após inserido o rato permaneceu vivo. Entretanto ,ao misturar a solução de bactéria CAP s morta com a bactéria CAP R viva o resultado encontrado não foi esperado, ou seja, o rato após um período foi à óbito. Portanto, haveria algo na solução da bactéria CAP S morta que transformava a bactéria CAP R viva em patogênica, assim como a CAP S viva. Sendo assim, em 1948 as pesquisadores Avery–MacLeod–McCarty deram continuidade a esse experimento e separaram a solução da bactéria CAP S morta por grupos representados por diferentes moléculas, como por exemplo um grupo de lipídeos, açucares e proteínas. E o resultado de óbito, ou seja, de transformação da bactéria CAP R viva em patogênica só ocorreu com a mistura da solução de ácidos nucleicos da bactéria CAP S morta. Assim, eles concluíram que os ácidos nucleicos (DNA) presentes no núcleo (material genético) são definitivamente os responsáveis por conter todas as informações genéticas das células.
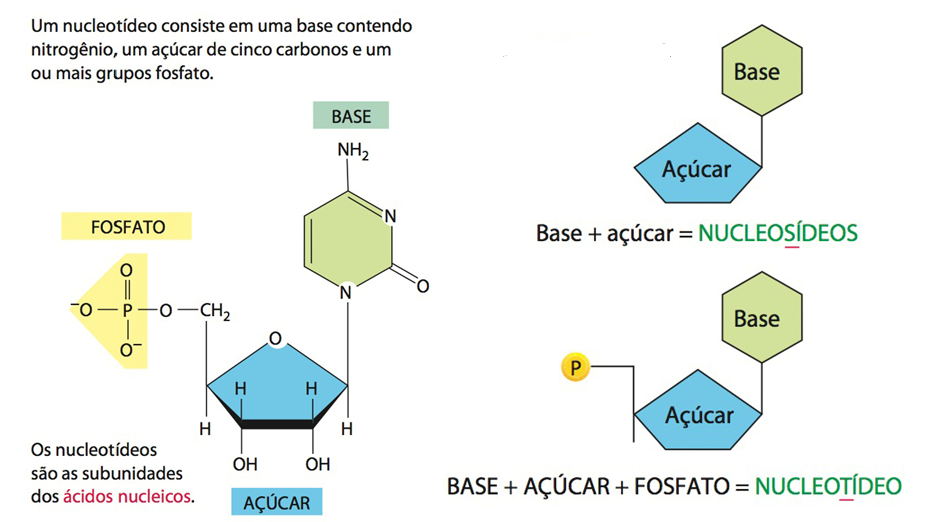
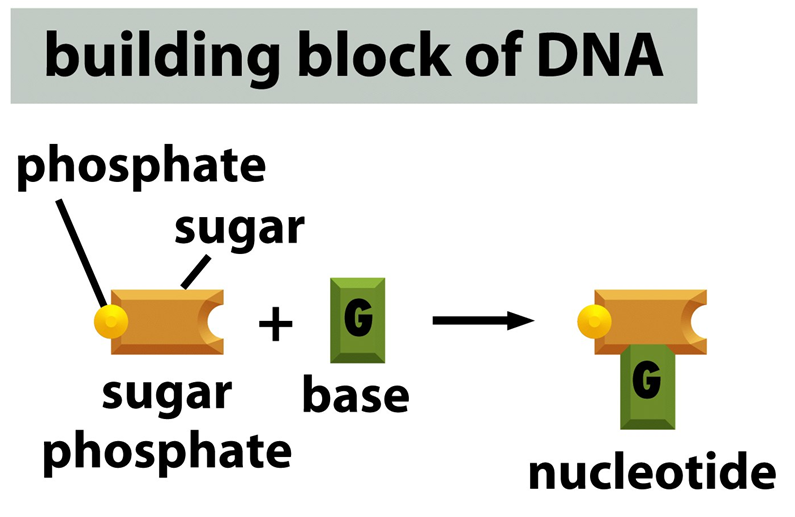
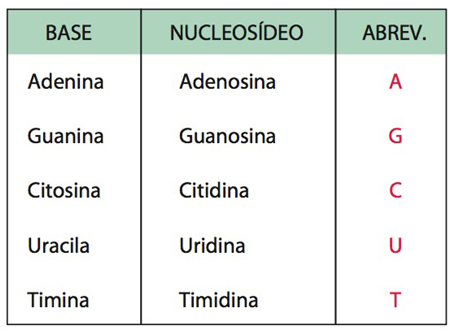
Fig. 5 Nucleotídeo e Nucleosídeo. Um nucleotídeo, como destacado na imagem, consiste em um açúcar que possui cinco carbonos (pentose), uma base contendo nitrogênio (base nitrogenada) ligada ao carbono 1´ do açucar e um ou mais grupos fosfatos ligado ao carbono 5´ do açúcar (A). Por conter grupos fosfatos, em alguns casos, são compostos que transportam muita energia, através das ligações dos mesmos, e portanto auxiliam nos processos metabólicos. Atuam também como sinais químicos, respondendo assim a hormônios e outros estímulos extracelulares. Entretanto, sua função mais significativa é de ser a base fundamental para a formação dos ácidos nucleicos, constituindo nosso material genético com todas nossas informações e hereditariedade. O Nucleosídeo é basicamente um Nucleotídeo que não possui o grupamento fosfato, como mostrado em B. São produtos de hidrólise química ou enzimática e ocorrem em quantidade muito pequena na célula.
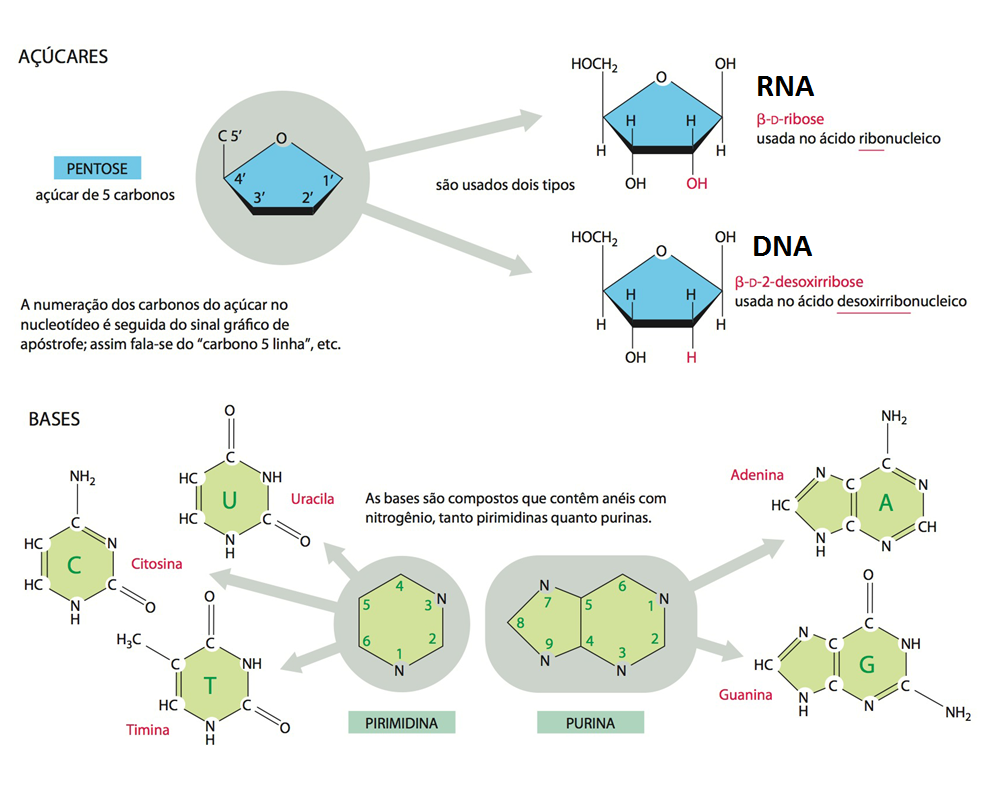
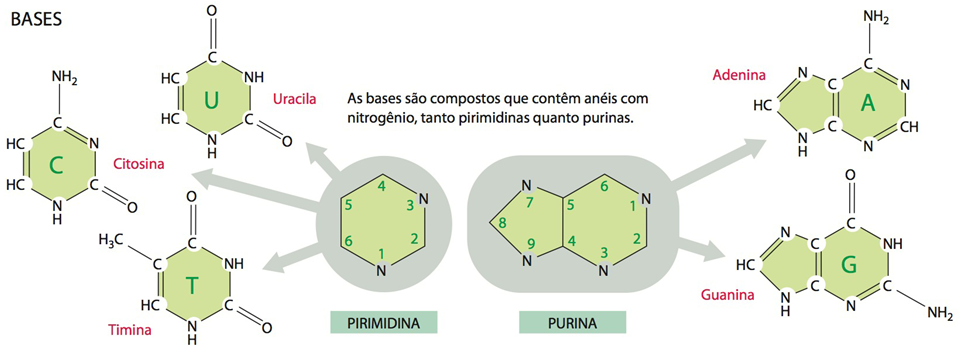
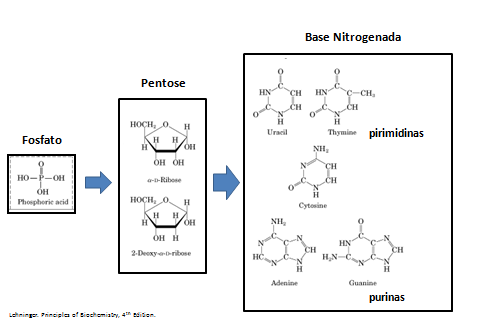
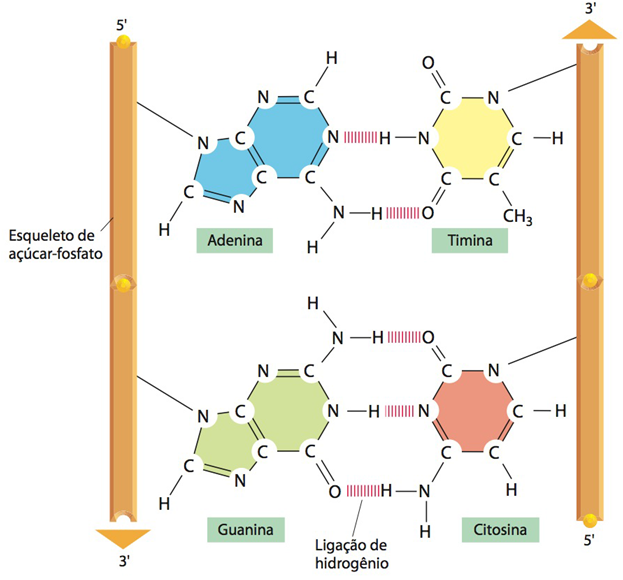
Fig. 6 Açucares e Bases. Como vimos, um nucleotídeo consiste em um açúcar que possui cinco carbonos, uma base contendo nitrogênio e um ou mais grupos fosfatos e é a subunidade dos ácidos nucleicos. Portanto, como uma estrutura com apenas três unidades pode conter toda a informação genética de uma célula e se diferenciar em DNA, RNA, ATP, GTP e tantas outras estruturas? A resposta está na diferenciação destas unidades como representado nas imagens acima. O RNA possui em seu açúcar um oxigênio a mais que o DNA, sendo assim, o RNA possui como açúcar a pentose chamada Ribose e o DNA possui a pentose chamada Desoxirribose. As bases se dividem em dois grupos: as pirimidinas, que são representadas por bases que contêm somente um anel de carbono com nitrogênio, a Uracila, Citosina e Timina; as purinas são representadas por bases que contêm dois anéis de carbono com nitrogênio, a Adenina e Guanina. Todos detalhes descritos anteriormente são facilmente identificados na imagem. No DNA estão presentes somente a Citosina que se une somente a Guanina e a Timina que se une somente com Adenina. No RNA estão presentes somente a Citosina, Guanina , Adenina e Uracila. As bases também variam em outras estruturas, como no ATP em que encontramos a Adenina e o GTP em que encontramos a Guanina.
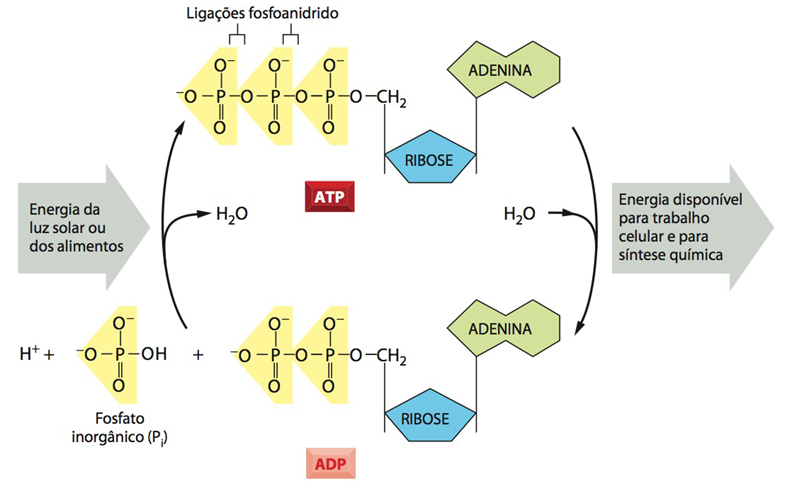
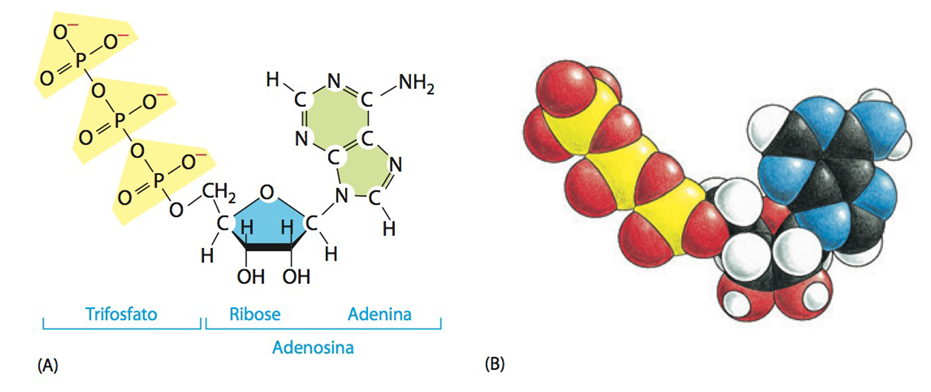
Fig.7 O ATP também serve como carreador de energia. Os nucleotídeos também podem carregar energia química por meio das ligações de alta energia entre seus grupamentos fosfato, chamadas “ligações anidrido fosfórico”, as quais são facilmente hidrolisáveis. A energia é liberada com a hidrólise (quebra com o uso de uma molécula de água) dessas ligações, liberando-se um fosfato inorgânico, um H+ e o nucleosídeo com os fosfatos restantes. A energia liberada pode ser usada para catalisar diferentes reações químicas. Os fosfatos estão normalmente ligados ao Carbono 5’ do glicídio (ribose ou desoxirribose) e conferem carga negativa ao nucleotídeo. De acordo com a quantidade de fosfatos, podem ser classificados como mono (AMP), di (ADP) ou trifosfato (ATP), conferindo carga negativa ao nucleotídeo. A base nitrogenada também pode variar (por exemplo, “ATP” para nucleotídeos com adenina e três fosfatos, ou “GDP” para nucleotídeos com guanina e dois fosfatos).
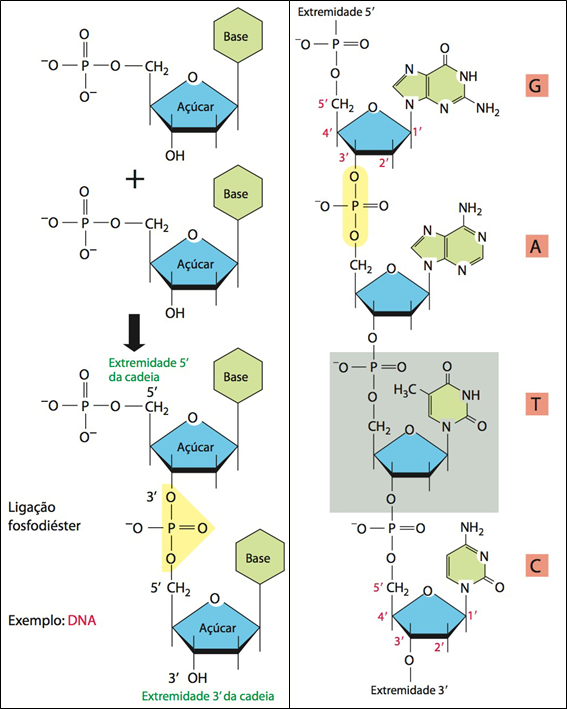
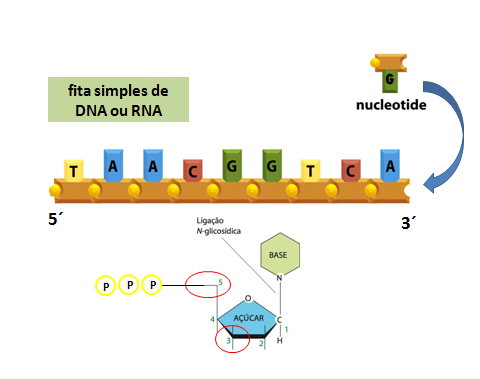
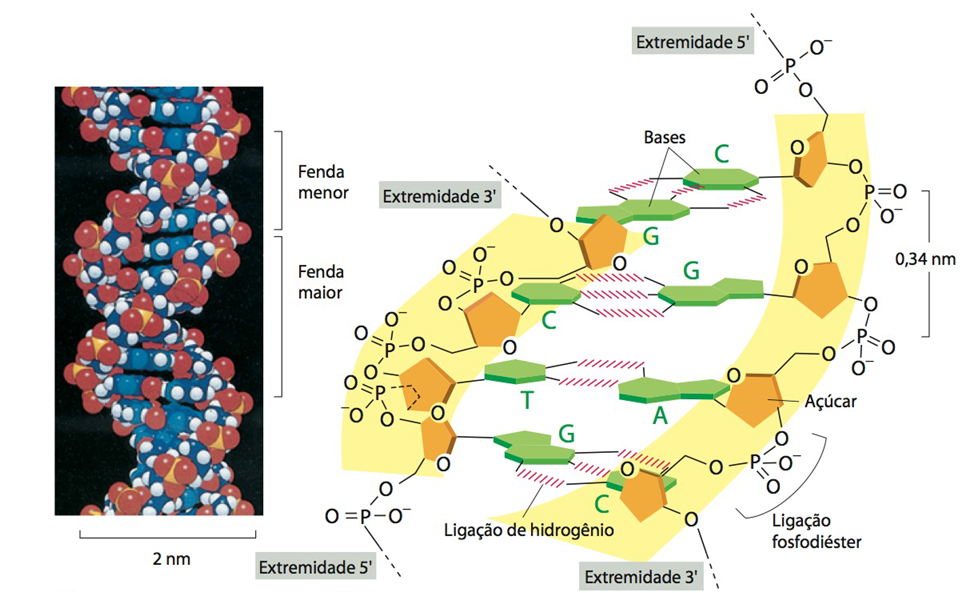
Fig.8 Ligação entre nucleotídeos: ligação fosfodiéster. O grupamento fosfatos está ligado ao Carbono 5’ do glicídio (ribose ou desoxirribose), enquanto o Carbono 3’ possui um grupamento hidroxila livre (3´-OH). Os nucleotídeos são ligados entre si por meio de “ligações fosfodiéster” entre o C5′ com o fosfato (de um nucleotídeo) e o C3′ com OH- (do nucleotídeo seguinte), formando uma sequência linear e garantindo polaridade à fita (uma extremidade 5’ e outra 3’). É devido à localização desses grupamentos livres que a polimerização do DNA ou RNA segue o sentido 5’ para 3’. Como cada nucleotídeo é constituído de uma base nitrogenada, a sequência dos nucleotídeos é nomeada de acordo com a base, podendo então ser A, T, C, G ou U. Ambas as figuras mostram a sequência de nucleotídeos usando como exemplo a desoxirribose; à direita estão evidenciadas as diferentes bases nitrogenadas.
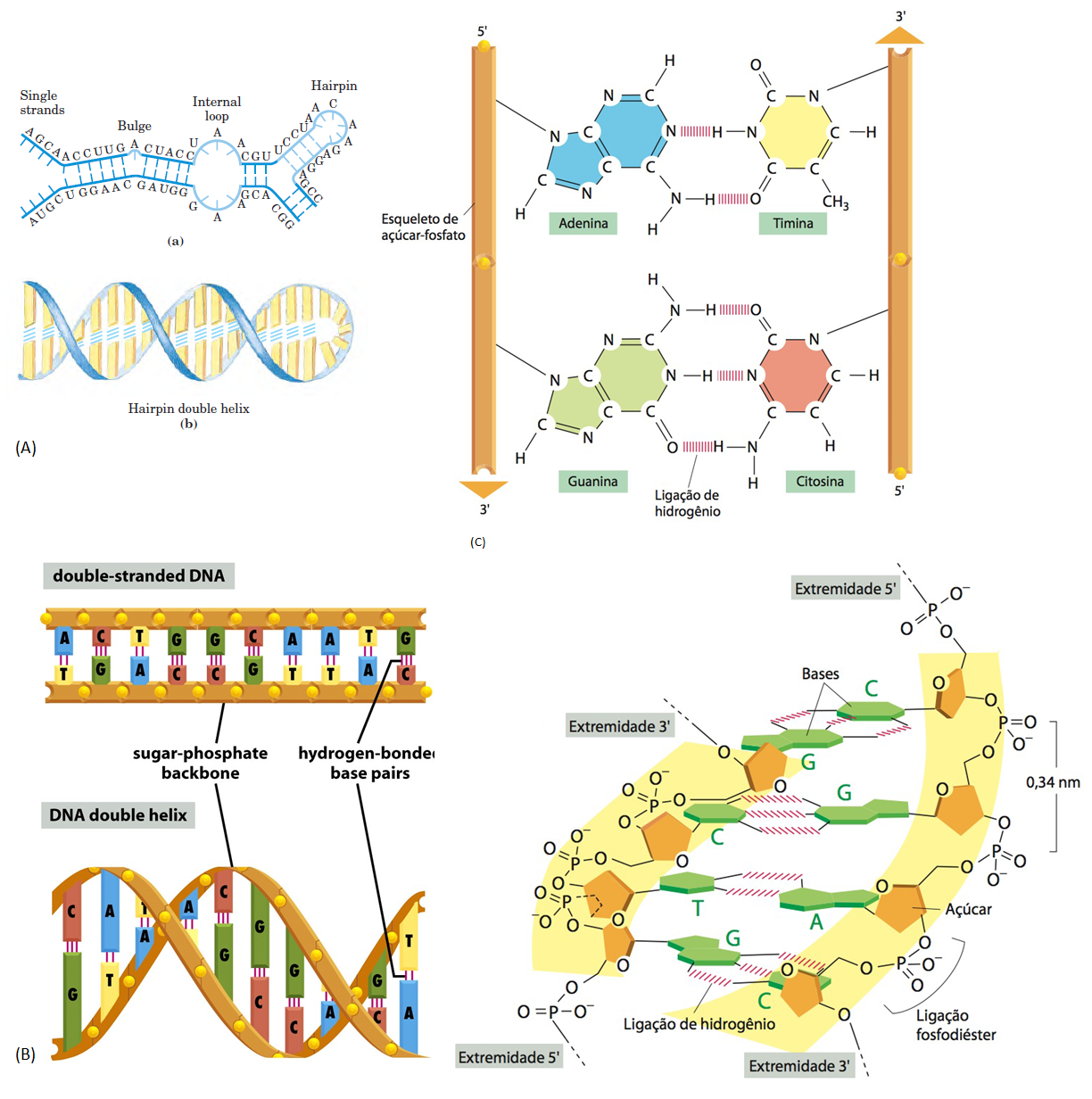
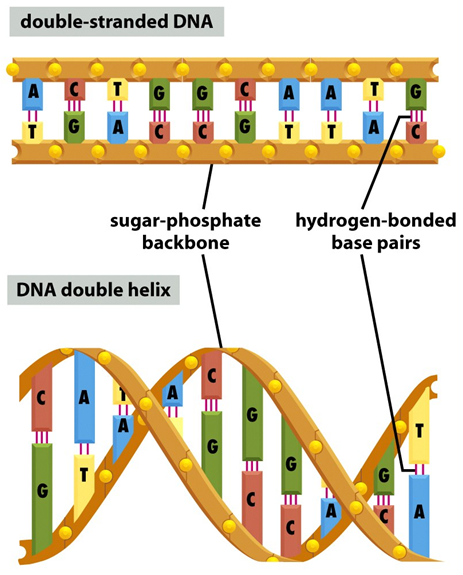
Fig.9 Estrutura do RNA e do DNA. O DNA (figura B) consiste em duas cadeias polipeptídicas compostas por quatro tipos de nucleotídeo, compostos pelo glicídio desoxirribose, sem OH- no Carbono 2’, um grupamento trifosfato e uma base nitrogenada, que podem ser guanina, adenina, citosina ou timina. Como visto na figura B, à direita, os nucleotídeos estão covalentemente ligados entre si através de fortes ligações fosfodiéster. As duas cadeias são complementares e antiparalelas (uma fita vai em direção oposta à sua fita complementar) e interagem e enrolando-se entre si, compactando-se e formando uma dupla-hélice. Enquanto as pentoses e fosfatos dos nucleotídeos unidos formam a parte externa da cadeia de DNA, ou o arcabouço (“coluna vertebral” ou “backbone”), as bases nitrogenadas permanecem na parte interna (perpendiculares ao arcabouço), entre as duas fitas; cada base liga-se à sua base complementar, da outra fita, por meio de ligações de hidrogênio, mais fracas do que as fosfodiéster: uma purina (G ou A) sempre se liga com uma pirimidina (C ou T); G sempre se liga com C (por meio de 3 ligações de Hidrogênio) e A, com T (2 ligações), devido à forma e estrutura química de suas bases (figura C). Os pares de bases mantêm distâncias similares, fazendo com que a distância entre as duas fitas seja sempre a mesma. As duas cadeias, então, enrolam-se uma ao redor da outra, compactando-se e formando a dupla-hélice. Já o RNA (figura A) é constituído por uma fita simples de nucleotídeos que pode interagir com si mesma e formar estruturas tridimensionais secundárias (por exemplo, o “hairpin”). O RNA tem como glicídio a ribose (possui uma hidroxila em 2’) e a fita é transcrita a partir de um molde de DNA, sendo que os nucleotídeos do RNA são complementares aos do molde; porém, ao invés de timina, a adenina do DNA pareia-se com uma uracila no RNA (a qual também é uma pirimidina). Na figura C também se encontra evidenciado o sentido de replicação da fita (de 5’ para 3’) pelas setas.
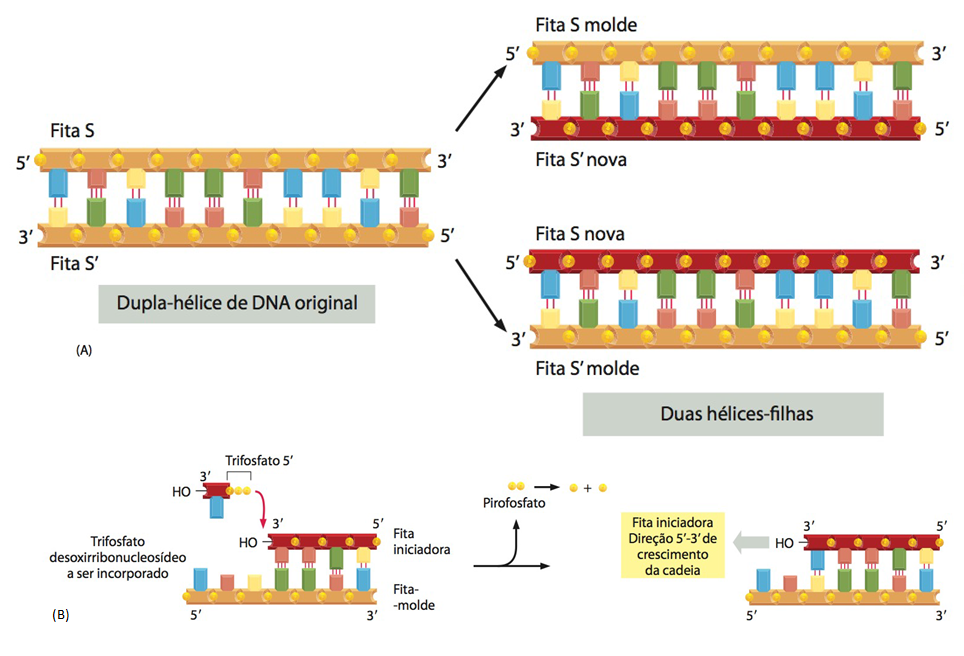
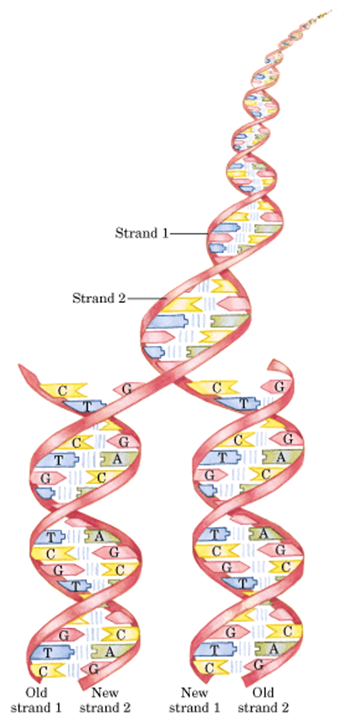
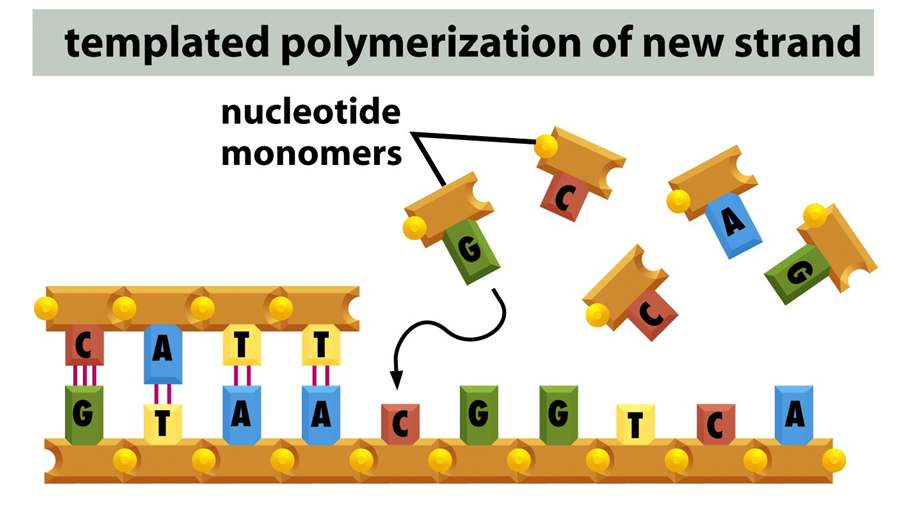
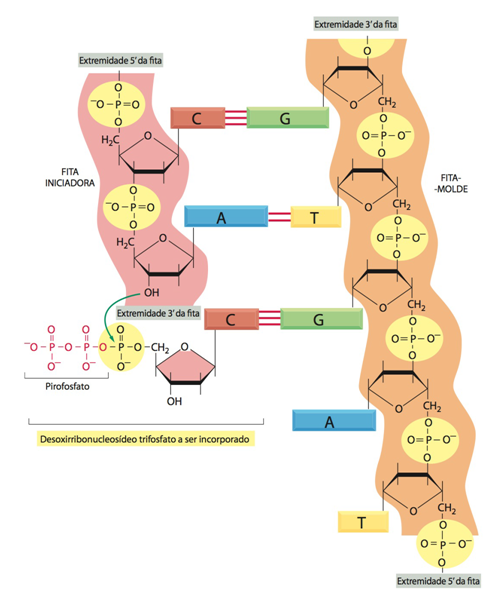
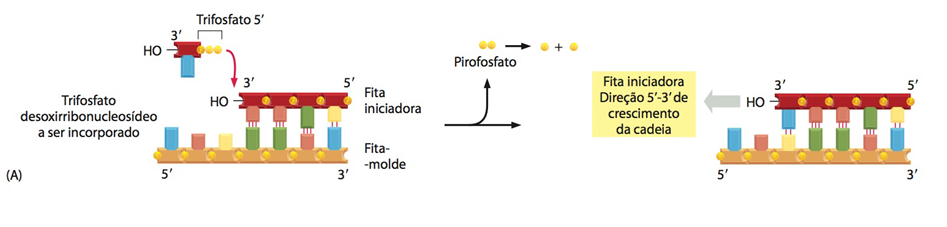
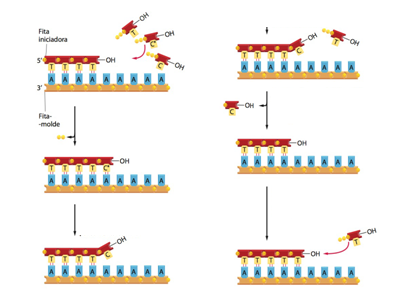
Fig.10 Pareamento de nucleotídeos e processo de replicação do DNA. A replicação do DNA é dita semiconservativa, já que a partir das duas fitas originais (da dupla fita despareada), as quais serão utilizadas como molde, é feita uma cópia precisa do DNA; uma cópia é feita a partir de cada fita-molde, logo, uma fita de DNA é capaz de gerar duas fitas-filhas. A fita original contém uma sequência de nucleotídeos exatamente complementar a sequência de sua fita original associada; cada fita atua como molde para a síntese de uma nova fita complementar. Isso permite que as sequências originais sejam conservadas nas duas fitas novas (ou fitas-filhas), e a informação genética seja passada para as células filhas. A figura B mostra o pareamento de um nucleotídeo a uma fita molde já em processo de replicação; o nucleotídeo perde dois grupamentos fosfatos (pirofosfato) na extremidade 5’ e liga-se na extremidade 3’ (com o grupamento OH-) do nucleotídeo já pareado à fita-molde, efetuando-se a ligação fosfodiéster. A energia utilizada para a formação da ligação entre os nucleotídeos vem da hidrólise e liberação do pirofosfato.
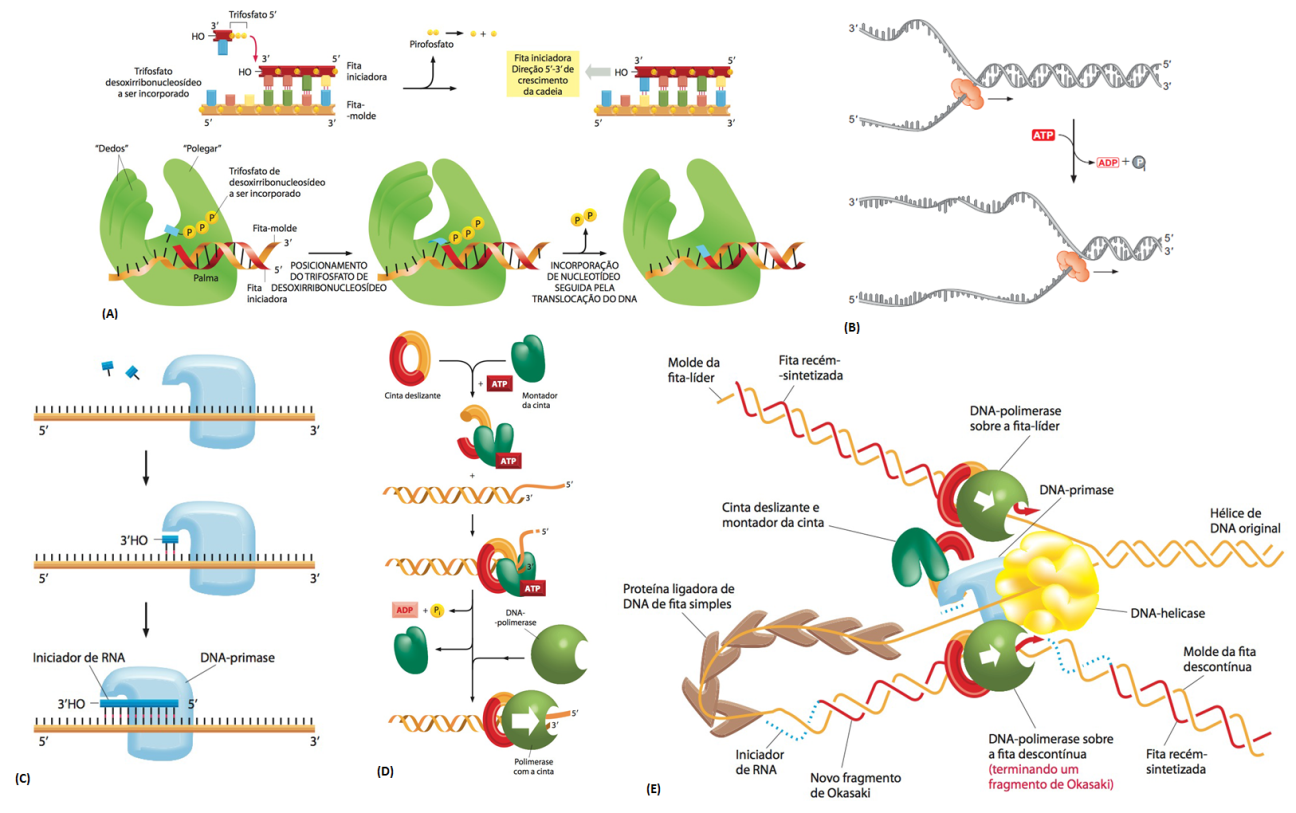
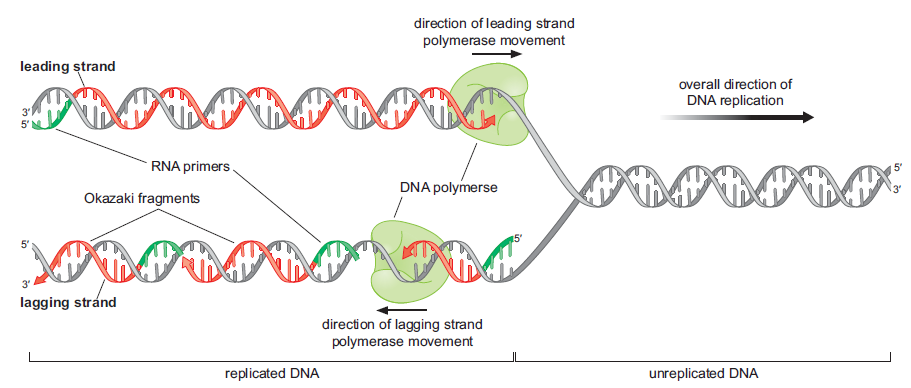
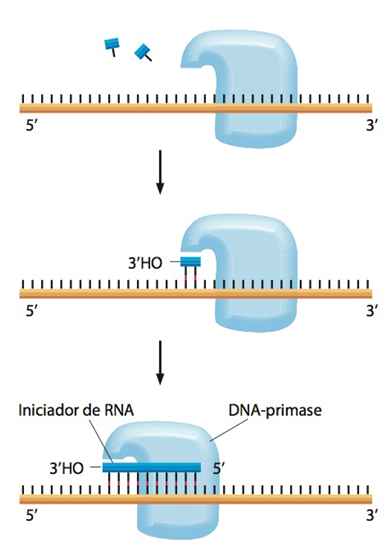
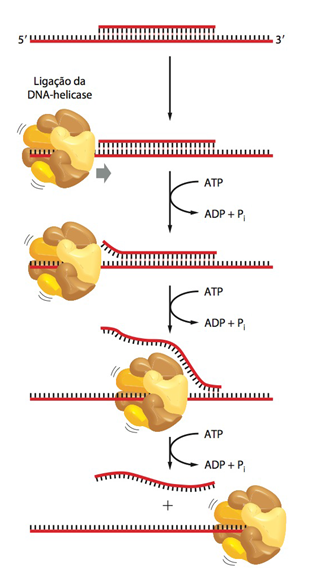
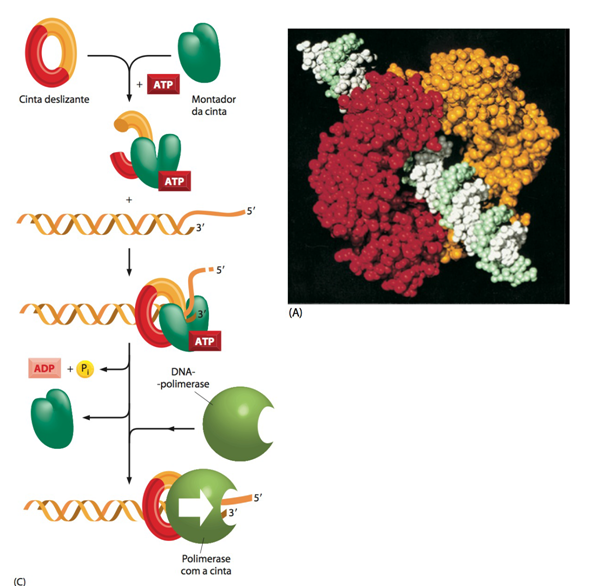
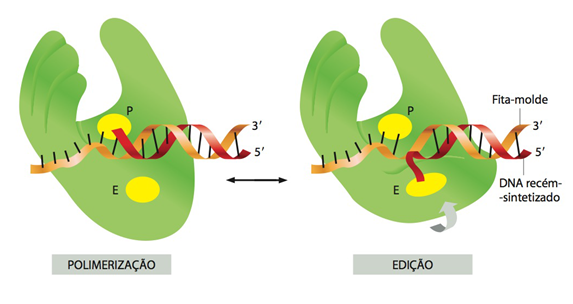
Fig.11 Início da replicação do DNA e principais enzimas envolvidas. O processo de replicação de DNA se inicia com a formação da forquilha de replicação e o despareamento da dupla fita (B). A DNA helicase se encarrega de desfazer a dupla-hélice nas fitas de DNA conforme “caminha” pela cadeia, formando a forquilha; proteínas SSB auxiliam a helicase, estabilizando a conformação e extensão da fita simples (B e E). Junto da forquilha estão DNA polimerases, enzimas que captam os trifosfatos de desoxirribonucleotídeo (dNTP) e fazem a polimerização do DNA (A). Cada fita usará uma DNA polimerase diferente e ambas atuam na direção 5′ – 3′ (A e E). Para que a DNA polimerase comece a atuar, a DNA primase polimeriza os primers de RNA com uma extremidade 3′ livre para que a replicação se inicie (C e E). Na fita contínua, apenas um primer é necessário, enquanto na descontínua é polimerizado um primer para cada fragmento de Okasaki (E). Os primers são removidos pela DNA polimerase e a DNA ligase conecta os fragmentos em ordem. A cinta deslizante mantém a DNA polimerase firmemente associada ao DNA enquanto esta “caminha” pela fita (D e E). Ela é formada por um complexo proteico, o montador de cinta (no caso de fragmentos de Okasaki, a cinta deslizante é desfeita e uma nova cinta é recolocada cada vez que um novo fragmento é sintetizado). Conforme a forquilha vai se deslocando na cadeia e separando a dupla-hélice, o DNA à frente fica super-enrolado, tensionando as forquilhas. Para desfazer essa tensão, as DNA topoisomerases, tipo I ou tipo II, formam suportes giratórios no DNA e clivam as fitas, permitindo que elas girem livremente, e depois as religam. A DNA polimerase (figura A) é a enzima que faz a polimerização dos trifosfatos de desoxiribonucleotídeos, formando a fita de DNA. Para que a replicação ocorra é necessário que o nucleotídeo já pareado à fita-molde (logo, a fita em formação) tenha a extremidade 3’ com OH- livre, possibilitando a formação da ligação fosfodiéster com o fosfato da extremidade 5’ dos nucleotídeos seguintes. Acima, a figura mostra que o nucleotídeo perde dois grupamentos fosfatos (pirofosfato) na extremidade 5’ e liga-se na extremidade 3’ (com o grupamento OH-) do nucleotídeo já pareado à fita-molde, efetuando-se a ligação fosfodiéster. A energia liberada pela hidrólise e liberação do pirofosfato é utilizada para fazer a ligação fosfodiéster entre os nucleotídeos. A DNA primase (figura C), polimeriza iniciadores na fita de DNA, os “primers de RNA”, que possuem a extremidade 3′ livre necessária para o início da replicação. O complexo enzimático chamado “montador de cinta” localiza-se na forquilha de replicação e forma as cintas deslizantes, hidrolisando ATP enquanto faz a montagem (no caso de fragmentos de Okasaki, a cinta deslizante é desfeita e uma nova cinta é recolocada cada vez que um novo fragmento é sintetizado). A DNA helicase (figura D) se encarrega de desfazer a dupla-hélice nas fitas de DNA conforme “caminha” pela cadeia, formando a forquilha de replicação, e é auxiliada por proteínas ligadoras de fita simples de DNA (SSB). A figura E mostra a posição dessas enzimas na forquilha.
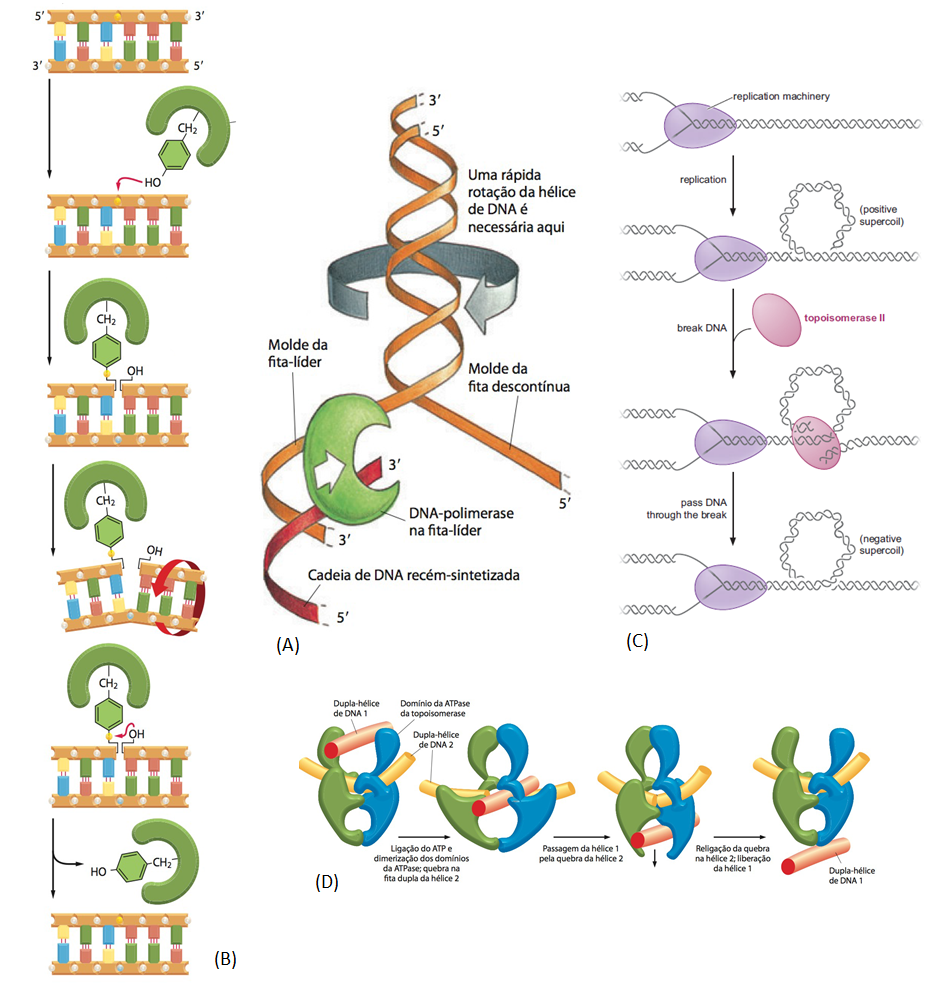
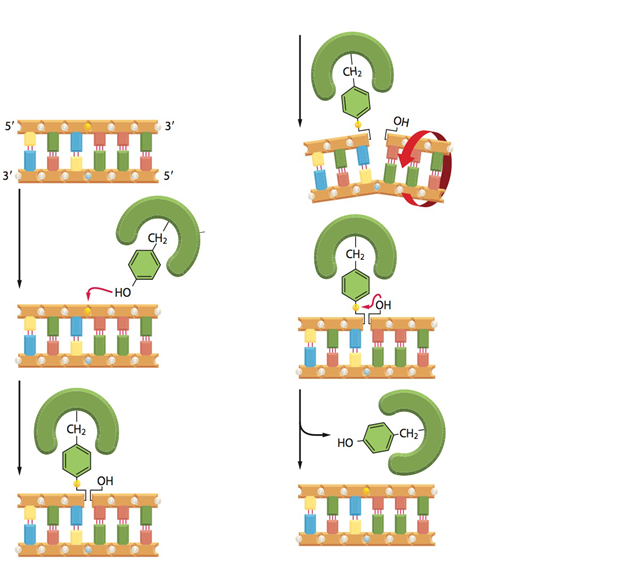
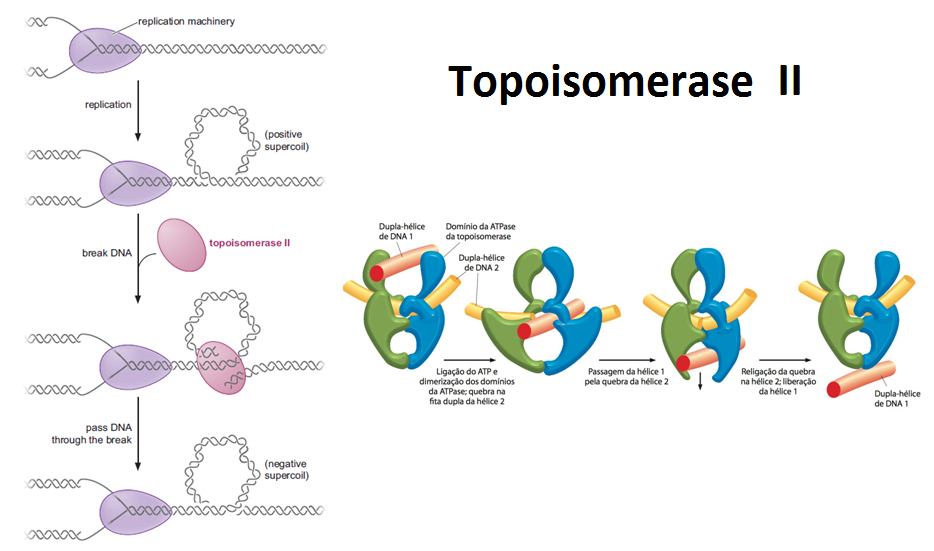
Fig.12 Enzima topoisomerase. Conforme a dupla fita de DNA é aberta pela enzima Helicase e a forquilha se desloca na fita , separando a dupla-hélice, o DNA à frente fica super-enrolado, tensionando as forquilhas (esquematizado na figura A). Para resolver esse problema, as enzimas DNA topoisomerases formam suportes giratórios no DNA. São dois tipos de topoisomerases: o tipo I (figura B) liga-se em uma fita simples e cliva-a, permitindo que as duas porções da fita (que permanece ligada) girem livremente, aliviando a tensão em pequenos segmentos da fita; posteriormente, a enzima refaz as ligações fosfodiéster entre os nucleotídeos, religando as duas porções clivadas. O tipo II (figura C e D) liga-se a duas duplas-hélices, clivando as duas fitas de uma delas ao mesmo tempo e quebrando sua dupla-hélice, permitindo a passagem da cadeia através dessa abertura, religando-a depois.
Imagens Complementares
Todas as imagens foram retiradas da seguinte fonte: (c) 2014 from Molecular Biology of the Cell, Sixth Edition by Alberts et al. Reproduced by permission of Garland Science/Taylor & Francis Group LLC.